Usando o AiNet
Acesse o Jupyter Notebook com o código disponível aqui!
Execute o notebook online via Binder:
Introdução
A clusterização é uma tarefa de aprendizado de máquina não supervisionado que visa agrupar um conjunto de dados no mesmo grupo (chamado de cluster).
Neste notebook, exploraremos o AiNet (Artificial Immune Network). O AiNet utiliza conceitos como afinidade de anticorpos e supressão de clones para identificar os centros dos clusters nos dados.
Objetivo: O objetivo é demonstrar a eficácia do AiNet na clusterização dos conjuntos de dados random:
- Blobs: Clusters bem definidos e esféricos.
- Moons: Clusters com uma forma não linear.
- Circles: Clusters dispostos em dois círculos concêntricos, apresentando separação não linear.
Estrutura do Notebook:
- Configuração: Instalação e importação das bibliotecas necessárias.
- Função de Visualização: Definição de uma função para plotar os resultados.
- Demonstração 1 - Dataset Blobs: Aplicação do AiNet ao blobs.
- Demonstração 2 - Dataset Moons: Aplicação do AiNet ao moons.
- Demonstração 3 - Dataset Circles: Aplicação do AiNet ao circles.
Importando a Rede Imune Artificial
from aisp.ina import AiNet
Função de Visualização
Função de Visualização do AiNet (plota agrupamentos e a rede imune)
def plot_immune_network(train_x, predict_y, model, title_prefix=""):
"""
Plota os resultados da clusterização da AiNet.
Paramentos:
train_x (np.array): Os dados de entrada.
predict_y (np.array): As previsões de cluster do modelo.
model (AiNet): O modelo AiNet treinado.
title_prefix (str, optional): Um prefixo para os títulos dos gráficos.
"""
clusters = list(model._memory_network.values())
network = np.array(model._population_antibodies)
_, axs = plt.subplots(2, 2, figsize=(12, 12))
colors = colormaps.get_cmap('Accent')
# Dados originais
axs[0][0].scatter(train_x[:, 0], train_x[:, 1], color='dodgerblue', alpha=0.9, s=50, marker='o', edgecolors='k')
axs[0][0].set_title(f'{title_prefix}Dados Originais', fontsize=16)
axs[0][0].set_xlabel('X', fontsize=14)
axs[0][0].set_ylabel('Y', fontsize=14)
axs[0][0].grid(True, linestyle='--', alpha=0.5)
# População de anticorpos
axs[0][1].scatter(network[:, 0], network[:, 1], color='crimson', alpha=0.9, s=70, marker='.', edgecolors='k')
axs[0][1].set_title(f'{title_prefix}População de Anticorpos', fontsize=16)
axs[0][1].set_xlabel('X', fontsize=14)
axs[0][1].set_ylabel('Y', fontsize=14)
axs[0][1].grid(True, linestyle='--', alpha=0.5)
# Previsões dos clusters
scatter = axs[1][0].scatter(train_x[:, 0], train_x[:, 1], c=predict_y, cmap='Accent', s=50, edgecolors='k', alpha=0.9)
axs[1][0].set_title(f'{title_prefix}Previsões dos Clusters (AiNet)', fontsize=16)
axs[1][0].set_xlabel('X', fontsize=14)
axs[1][0].set_ylabel('Y', fontsize=14)
axs[1][0].grid(True, linestyle='--', alpha=0.5)
legend1 = axs[1][0].legend(*scatter.legend_elements(), title="Clusters")
axs[1][0].add_artist(legend1)
# Grafo da Rede Imune
G = nx.Graph()
positions = {}
for i, cluster in enumerate(clusters):
cluster_nodes = [f'{i}_{j}' for j in range(len(cluster))]
G.add_nodes_from(cluster_nodes)
for node, point in zip(cluster_nodes, cluster):
positions[node] = tuple(point)
dist_matrix = squareform(pdist(cluster))
mst_local = minimum_spanning_tree(dist_matrix).toarray()
for row_idx, row in enumerate(mst_local):
for col_idx, weight in enumerate(row):
if weight > 0:
G.add_edge(cluster_nodes[row_idx], cluster_nodes[col_idx], weight=weight)
for i, cluster in enumerate(clusters):
cluster_nodes = [f'{i}_{j}' for j in range(len(cluster))]
nx.draw_networkx_nodes(G, positions, nodelist=cluster_nodes, ax=axs[1][1],
node_color=[colors(i)], node_size=70, edgecolors='k', label=f'Cluster {i}')
nx.draw_networkx_edges(G, positions, ax=axs[1][1], alpha=0.6)
axs[1][1].set_title(f'{title_prefix}Rede Imune Grafo', fontsize=16)
axs[1][1].set_xlabel('X', fontsize=14)
axs[1][1].set_ylabel('Y', fontsize=14)
axs[1][1].grid(True, linestyle='--', alpha=0.5)
axs[1][1].legend()
plt.tight_layout()
plt.show()
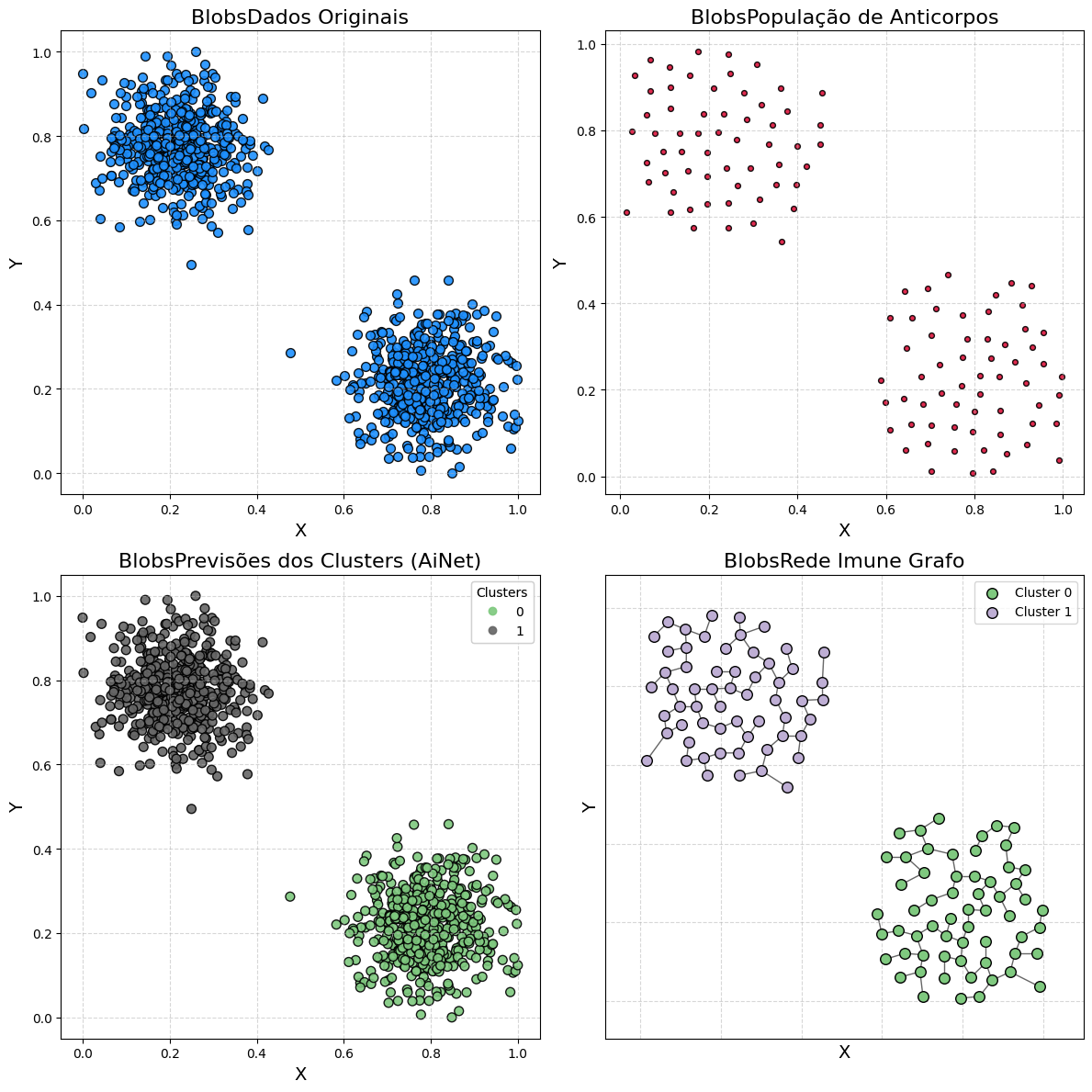
Demonstração 1 - Dataset Blobs
Gerando bolhas de dados
samples, output = make_blobs(
n_samples=1000,
cluster_std=0.07,
center_box=(0.0, 1.0),
centers=[[0.25, 0.75], [0.75, 0.25]],
random_state=1234,
)
Treinando o modelo AiNet
model = AiNet(suppression_threshold=0.96, affinity_threshold=0.95, mst_inconsistency_factor=3, seed=123)
predict_y = model.fit_predict(samples)
Output:
✔ Set of memory antibodies for classes (0, 1) successfully generated | Clusters: 2 | Population of antibodies size: 119: ┇██████████┇ 10/10 total training interactions
Silhouette score
silhouette = silhouette_score(samples, predict_y)
print(f"Coeficiente de Silhueta: {silhouette:.3f}")
Output:
Coeficiente de Silhueta: 0.826
Visualizando os resultados
plot_immune_network(samples, predict_y, model, title_prefix="Blobs - ")
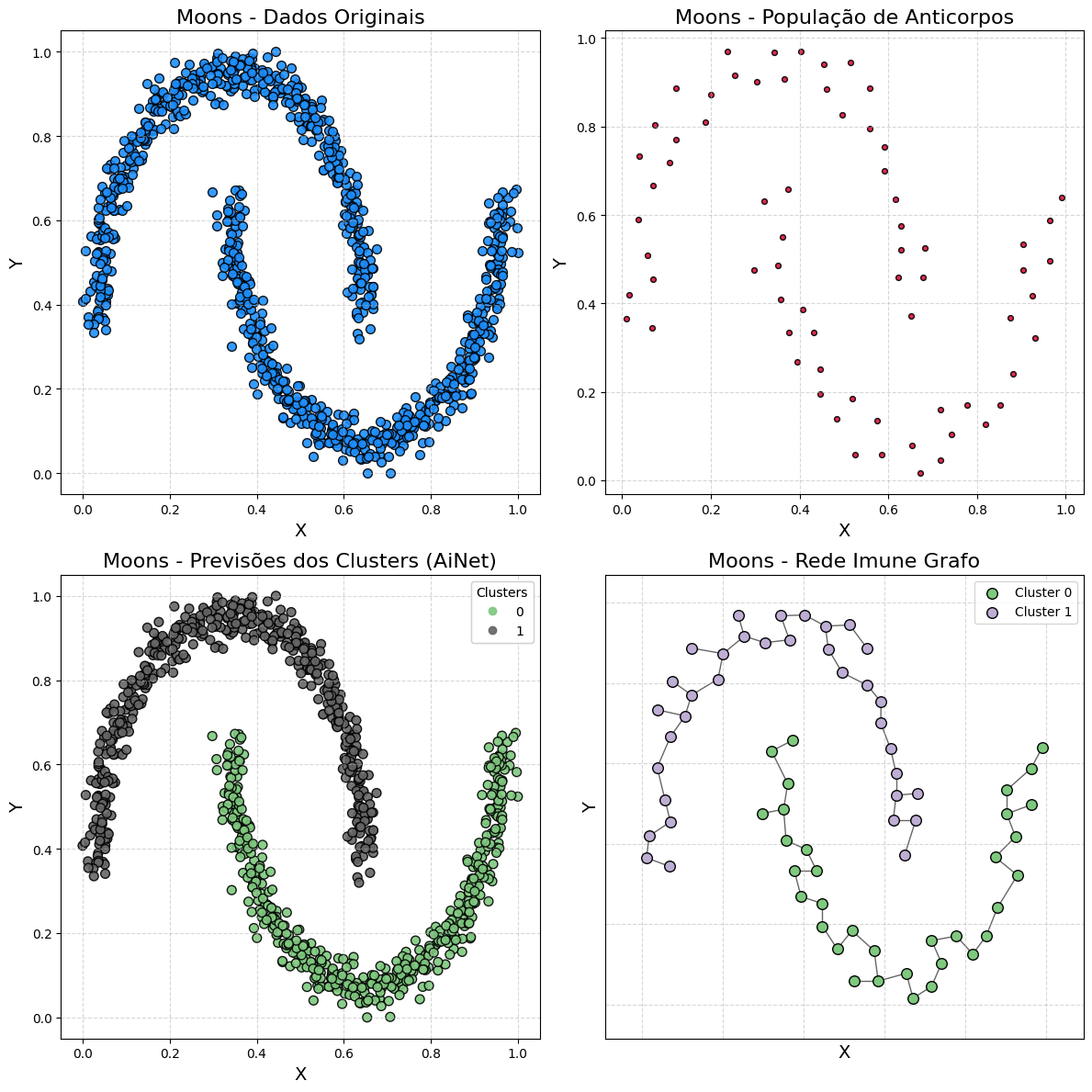
Demonstração 2 - Moons Dataset
Gerando dados em formato de "moons" (luas)
samples, output = make_moons(n_samples=1000, noise=0.05, random_state=42)
samples = MinMaxScaler().fit_transform(samples)
Treinando o AiNet
model = AiNet(suppression_threshold=0.95, affinity_threshold=0.97, mst_inconsistency_factor=2.5, seed=123)
predict_y = model.fit_predict(samples)
Output:
✔ Set of memory antibodies for classes (0, 1) successfully generated | Clusters: 2 | Population of antibodies size: 69: ┇██████████┇ 10/10 total training interactions
Silhouette score
silhouette = silhouette_score(samples, predict_y)
print(f"Coeficiente de Silhueta: {silhouette:.3f}")
Output:
Coeficiente de Silhueta: 0.398
Visualizando
plot_immune_network(samples, predict_y, model, title_prefix="Moons - ")
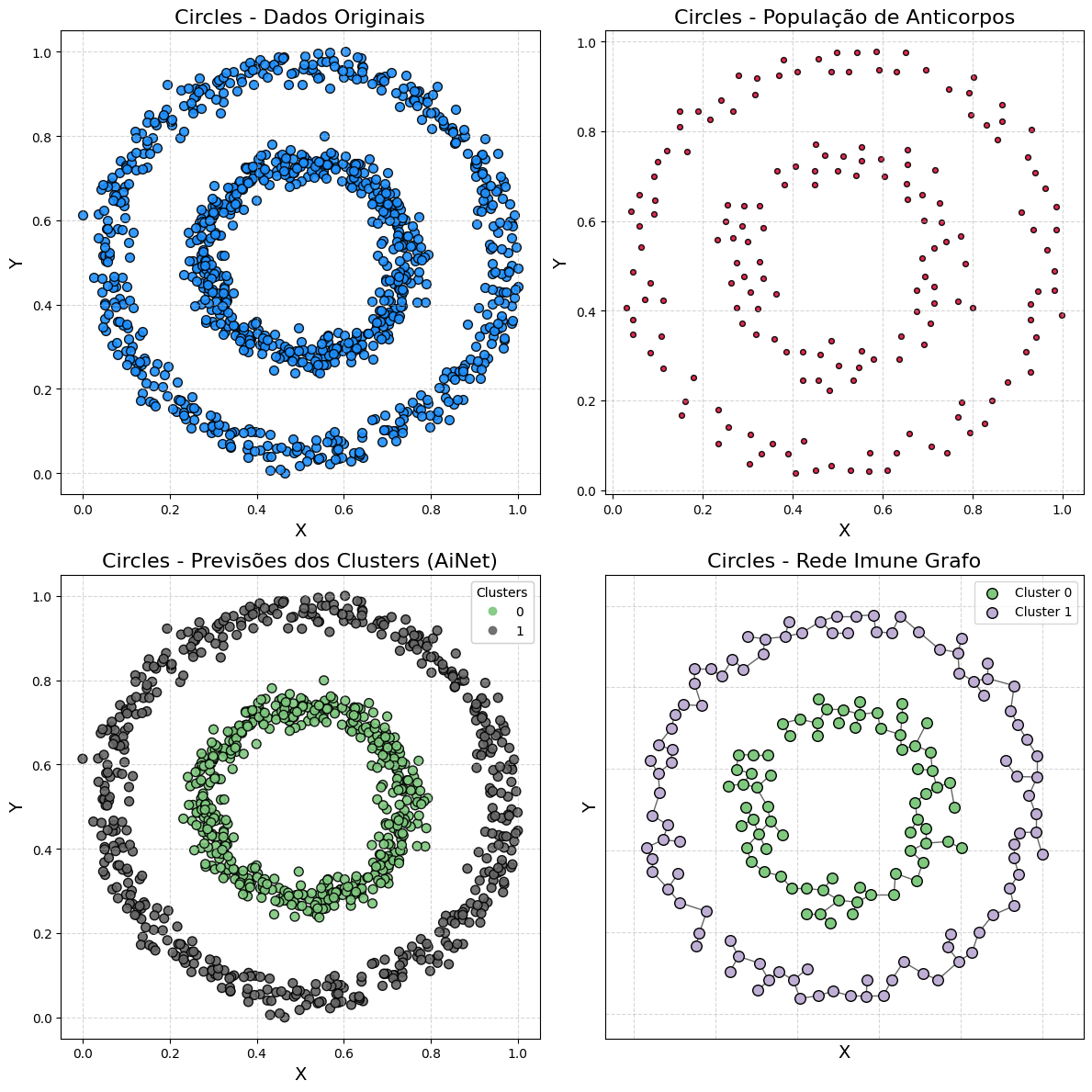
Demonstração 3 - Circles Dataset
Gerando dados em formato de círculos concêntricos
samples, output = make_circles(n_samples=1000, noise=0.05, factor=0.5, random_state=42)
samples = MinMaxScaler().fit_transform(samples)
Training AiNet
model = AiNet(suppression_threshold=0.97, affinity_threshold=0.98, mst_inconsistency_factor=3.8, seed=123)
predict_y = model.fit_predict(samples)
Output:
✔ Set of memory antibodies for classes (0, 1) successfully generated | Clusters: 2 | Population of antibodies size: 169: ┇██████████┇ 10/10 total training interactions
Silhouette score
silhouette = silhouette_score(samples, predict_y)
print(f"Coeficiente de Silhueta: {silhouette:.3f}")
Output:
Coeficiente de Silhueta: 0.112
Visualizando o AiNet nos dados de círculos
plot_immune_network(samples, predict_y, model, title_prefix="Circles - ")